近日,我院戈惠明教授與焦瑞華教授團隊在J. Am. Chem. Soc.上刊發了基因組挖掘P450酶修飾RiPPs的最新成果。該研究通過從頭編寫前體肽基因的識别程序并組建一套簡便高效的基因組挖掘流程,一次性揭示了細菌中P450酶修飾RiPPs的全局性分布藍圖,基于此,通過異源表達,從4個不同類别中鑒定了11個新穎的P450酶修飾RiPPs。
核糖體合成和翻譯後修飾肽(RiPPs)是一類結構與活性十分多樣的天然産物,其生物合成始于蛋白質翻譯過程生成的前體肽,再經後續修飾酶的加工,形成終産物。在衆多的後修飾類型中,環化最為普遍,可以顯著增強多肽産物的生物活性和結構上的剛性(圖1A)。
近年來,P450酶被報道可以催化前體肽中芳香氨基酸側鍊間的交聯成環(圖1B, C),形成的環肽産物稱之為P450酶修飾RiPPs。為了進一步拓展該類環肽分子的化學多樣性,該團隊利用生物信息學手段對細菌展開了基因組挖掘。由于P450酶數量衆多,且前體肽基因難以預測,該團隊從頭建立了一個簡便高效的基因組挖掘流程(圖1D),從UniRef90數據庫中收集了76,988條P450酶基因及上下遊400bp的非編碼區序列,利用預測程序,獲得了12,847條短肽序列(圖1D)。再以短肽序列C端區域是否富含芳香氨基酸,從中篩選到2,648個對應的P450酶,通過這些P450酶構築的序列相似性網絡和對應的前體肽序列保守性分析,該團隊一共發現了16個P450酶修飾RiPPs的不同類别(圖2,類别A-P),較為完整的揭示了該類環肽産物在細菌中的全局性分布。
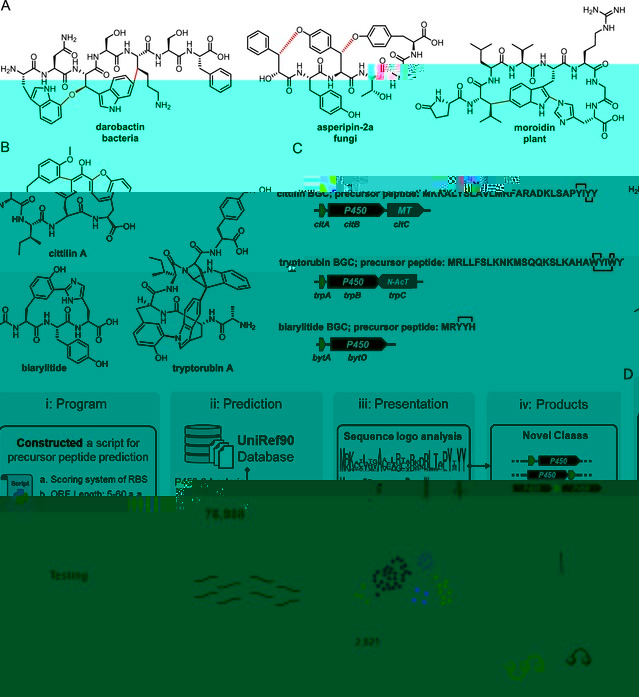
圖1 芳香氨基酸側鍊交聯的RiPPs和P450酶修飾RiPPs的挖掘流程

圖2 P450酶修飾RiPPs在細菌中的全局性分布和前體肽的序列保守性分析
随後,該團隊以全局性分布藍圖為指導,開展結構表征工作,以期獲得結構類型多樣的新穎環肽分子。通過在鍊黴菌S. albus J1074和大腸杆菌中的異源表達,以及後續的分離純化和結構鑒定,共計獲得了11個新穎的環肽分子(圖3)。同時,通過體外酶促反應實驗,證實了環肽化合物8生物合成基因簇中P450酶的功能。

圖3 本研究中發現的新穎環肽分子
總而言之,該研究通過從頭編寫前體肽基因的識别程序并組建一套簡便高效的基因組挖掘流程,描繪了P450酶修飾RiPPs的全局藍圖,并通過多個新穎環肽産物的表征,證實了該類環肽産物巨大的結構多樣性潛能。這項研究為環肽天然産物的發現提供了新的視角,并為環肽藥物的人工合成提供了新的思路和酶學工具。
伟徳国际官网登录入口博士後胡逸靈、博士生殷方洲為文章的共同第一作者,伟徳国际官网登录入口戈惠明教授、焦瑞華教授為文章的通訊作者。該研究得到了科技部、國家自然科學基金委員會和海南省重點研發計劃等項目的資助。
文章題目:P450-Modified Ribosomally Synthesized Peptides with Aromatic Cross-Links
全文鍊接:https://doi.org/10.1021/jacs.3c07416